Content from Datenstrukturen
Zuletzt aktualisiert am 2026-02-24 | Diese Seite bearbeiten
Geschätzte Zeit: 20 Minuten
Übersicht
Fragen
- Welche “Datenarten” gibt es?
- Wie organisiere ich tabellarische Daten?
- Was ist “tidy”?
Ziele
- Standarddatentypen in R
-
data.frameundtibble - “tidy” Konzept
Für die Verarbeitung von tabellarischen Daten werden in R i.d.R.
data.frames verwendet. Das tidyverse package
liefert eine erweiterte Version des data.frames, das
tibble, welches etwas transparenter in seiner Verwendung
ist. In einem tibble (oder data.frame)
entspricht jede Zeile einem Datensatz (oder einer Beobachtung) und jede
Spalte einer Variable (oder einem Merkmal) dieses Datensatzes. Damit
haben alle Daten in der gleichen Spalte den gleichen Datentyp, z.B.
“Zahl” (numeric), “Kommazahl” (double),
“Ganzzahl” (integer), “Text” (character),
“Datum” (date), etc. Im Folgenden wird der Beispieldatensatz
storms aus dem dplyr package gezeigt.
R
dplyr::storms
AUSGABE
# A tibble: 20,778 × 13
name year month day hour lat long status category wind pressure
<chr> <dbl> <dbl> <int> <dbl> <dbl> <dbl> <fct> <dbl> <int> <int>
1 Amy 1975 6 27 0 27.5 -79 tropical d… NA 25 1013
2 Amy 1975 6 27 6 28.5 -79 tropical d… NA 25 1013
3 Amy 1975 6 27 12 29.5 -79 tropical d… NA 25 1013
4 Amy 1975 6 27 18 30.5 -79 tropical d… NA 25 1013
5 Amy 1975 6 28 0 31.5 -78.8 tropical d… NA 25 1012
6 Amy 1975 6 28 6 32.4 -78.7 tropical d… NA 25 1012
7 Amy 1975 6 28 12 33.3 -78 tropical d… NA 25 1011
8 Amy 1975 6 28 18 34 -77 tropical d… NA 30 1006
9 Amy 1975 6 29 0 34.4 -75.8 tropical s… NA 35 1004
10 Amy 1975 6 29 6 34 -74.8 tropical s… NA 40 1002
# ℹ 20,768 more rows
# ℹ 2 more variables: tropicalstorm_force_diameter <int>,
# hurricane_force_diameter <int>Bei der Verwendung von tibbles sind unter den
Spaltennamen die Datentypen der Spalten abgekürzt dargestellt.
data.frames oder tibbles können auch aus
anderen Datenstrukturen erstellt werden, z.B. aus vectors,
lists oder matrices. Kleinere Datensätze
können auch direkt als tibble erstellt werden, indem die
Daten in die Funktion tibble() eingegeben werden.
R
marriages <-
tibble(
name = c("Alice", "Bob", "Charlie","Diana"),
age = c(25, 30, 35, 12),
married = c(TRUE, FALSE, TRUE, FALSE)
)
Ziel der Datenverarbeitung ist es, die Daten so zu transformieren, dass sie für die Analyse und Visualisierung geeignet sind und ein Format wie oben gezeigt vorweisen. Wenn das der Fall ist, sprich
- jede Zeile entspricht einem Datensatz und
- jede Spalte entspricht einer Variable,
wird der Datensatz als “tidy” bezeichnet, was im Folgenden visualisiert wird.

Spaltenzugriff
Betrachten sie den Datensatz marriages von oben.
Wie können Sie nur die Spalte name aus dem Datensatz
extrahieren?
R
# base R
marriages$name # mit "$" ohne Anführungszeichen (Standardfall)
marriages$`name` # mit "$" und "backtick" quotes für Variablennamen mit Sonderzeichen
marriages[["name"]] # WICHTIG: Doppelte eckige Klammern und Name in quotes, siehe unten !
marriages[[1]] # mit doppelter eckiger Klammer und Spaltenindex (nicht empfohlen, sicherer via Namen)
# tidyverse dplyr package
dplyr::pull(marriages, name) # ohne "dplyr::" wenn dplyr geladen ist (library(dplyr))
# oder in einer pipe, was später diskutiert wird
marriages %>% dplyr::pull(name)
Achtung: - EINFACHE eckige Klammern marriages["name"]
geben ein data.frame zurück (REDUZIEREN die
aktuelle Datenstruktur), während - DOPPELTE eckige Klammern
marriages[["name"]] den vector der Spalte
name zurückgeben (also das benannte Element
EXTRAHIEREN)!
Datentypen
Welche Datentypen haben die Spalten des Datensatzes?
-
name:character -
age:numericalso generell “Zahlen”- entspricht i.d.R.
doubleist eine Unterart vonnumeric, die auch Kommazahlen enthält. -
integerwäre auch möglich, abernumericist der Standarddatentyp für Zahlen in R. - wenn wir explizit Ganzzahlen definieren wollen, müssen
diese mit einem “L” versehen werden, z.B.
25L, oder die Spalte explizit alsintegerdefinier/umgewandelt werden, z.B.as.integer(c(25, 30, 35)).
- entspricht i.d.R.
-
married:logical
Aufbau
Wieviele Beobachtungen und Variablen hat der Datensatz?
- Beobachtungen: 4 = Anzahl Zeilen (oder Länge der Spalten)
- Variablen: 3 = Anzahl Spalten
Um zu verdeutlichen, worin der Unterschied zwischen
data.frame und tibble besteht, führen sie
folgende Code in einer R-Session aus:
R
# Erstellen eines data.frame
data.frame( "das sind daten" = c(1, 2, 3), "das sind auch daten" = c(4, 5, 6) )
# Erstellen eines äquivalenten tibbles
tibble::tibble( "das sind daten" = c(1, 2, 3), "das sind auch daten" = c(4, 5, 6) )
Unterschiede
Was fällt ihnen auf?
AUSGABE
das.sind.daten das.sind.auch.daten
1 1 4
2 2 5
3 3 6AUSGABE
# A tibble: 3 × 2
`das sind daten` `das sind auch daten`
<dbl> <dbl>
1 1 4
2 2 5
3 3 6- Spaltennamen in
data.framewerden automatisch bereinigt, damit sie den normalen Konventionen für Variablennamen entsprechen, z.B. Leerzeichen werden durch Punkte ersetzt. -
tibblezeigt die Spaltennamen so an, wie sie eingegeben wurden, und unterstützen auch Sonderzeichen in den Spaltennamen.- beachten Sie, dass Sonderzeichen in Spaltennamen in R nicht empfohlen werden, da sie zu Problemen führen können.
- ausserdem müssen solche Spaltennamen später in der Verwendung in backtick “`” quotes gesetzt werden, siehe oben
- ausserdem (hier nicht zu sehen) zeigt
tibblenur die ersten 10 Zeilen an, währenddata.framealle Zeilen anzeigt. - zudem werden die Datentypen für jede Spalte in
tibbleangezeigt, was beidata.framenicht der Fall ist.
- Spalten in einem
tibblewerden auch Variablen (des Datensatzes) genannt.- Eine Spalte ist ein
vector, d.h. es können nur Werte des gleichen Datentyps enthalten sein.
- Eine Spalte ist ein
- Zeilen in einem
tibblewerden auch Beobachtungen (des Datensatzes) genannt. - Ein Datensatz ist “tidy”,
- wenn jede Zeile einem Datensatz und jede Spalte einer Variable entspricht.
- Vereinfacht: wenn man beim Visualisieren der Daten nur jeweils eine Zeile pro Datenpunkt benötigt und keine doppelt verwendet wird.
Dieses Dokument wurde mit Unterstützung von GitHub Copilot erstellt, einem KI-gestützten Autocompletion-Tool, das auf der OpenAI GPT-3-Technologie basiert.
Content from Datenimport aus Dateien
Zuletzt aktualisiert am 2026-02-24 | Diese Seite bearbeiten
Geschätzte Zeit: 30 Minuten
Übersicht
Fragen
- Wie importiere ich Daten? (
readr,readxl, …) - Auf was muss ich achten?
Ziele
- Datenimport via
readrundreadxl - Kodierung von Zahlen und Texten
- Pfadangaben
In R können Daten aus verschiedenen Quellen und von verschiedenen Orten eingelesen werden.
Die gängigsten Quellen sind:
- Textdateien (z.B.
.csv,.tsv,.txt) - Excel-Dateien (z.B.
.xlsx,.xls) - Datenbanken (z.B.
MySQL,PostgreSQL,SQLite)
Die gängigsten Orte sind:
- lokale Dateien
- URLs, also direkter Datenimport aus dem Internet oder lokalem Netzwerk
Im Folgenden einige Beispiele zum Einlesen von Daten aus Dateien in
R. Die lokalen Dateien müssen sich im Arbeitsverzeichnis
befinden, das mit getwd() abgefragt und mit
setwd() gesetzt werden kann (oder mit entsprechenden
Menüeinträgen in RStudio), oder mit vollem Verzeichnisinformationen
angegeben werden (nicht gezeigt und nicht empfohlen). Um den folgenden
Beispielcode mit lokalen Dateien auszuführen, müssen daher die Dateien
storms-2019-2021.csv und storms-2019-2021.xlsx erst ins
aktuelle Arbeitsverzeichnis gedownloaded werden.
R
library(readr)
# CSV aus dem Internet
read_csv("https://raw.githubusercontent.com/tidyverse/dplyr/master/data-raw/storms.csv")
# lokale Excel-Datei
readxl::read_xlsx("storms-2019-2021.xlsx",
sheet = "storms-2020") # explizite Angabe des Tabellenblatts
# lokale CSV Datei Semikolon getrennt (";") und mit westeuropäischem Zahlenformat ("," als Dezimaltrenner)
read_csv2("storms-2019-2021.csv")
# explizites Einstellen des Daten- und Dateiformats
# (hier analog zu den Standardeinstellungen von `read_csv2()`)
read_delim(
"storms-2019-2021.csv", # Dateiname
delim = ";", # Spaltentrenner der Datei
locale = locale( # zusätzliche Einstellungen wie gespeicherte Informationen kodiert sind
decimal_mark = ",", # Dezimaltrenner
grouping_mark = ".", # Tausendertrenner
encoding = "latin1" # (alte) westeuropäisches Buchstabenkodierung (z.B. Umlaute)
)
)
Beachten sie im letzten Beispiel das explizite Setzen des
Textencodings, um sicherzustellen, dass Umlaute korrekt eingelesen
werden (hier im Beispiel wird latin1 verwendet, was für
westeuropäische Länder wie Deutschland oder Frankreich üblich ist bzw.
war). Dies ist vor allem beim Import von alten Daten wichtig,
die in einem anderen Zeichensatz kodiert sein könnten. Heutzutage wird
meist UTF-8 verwendet, was ein internationaler Standard ist
und alle Zeichen korrekt darstellt. Dies ist auch das Standardencoding
für die meisten Funktionen wie z.B. in readr und
readxl.
Zudem wird im obigen read_delim() Beispiel die
Zahlenkodierung explizit gesetzt. Das heisst, dass Dezimaltrenner und
Tausendertrenner explizit angegeben werden, da z.B. westeuropäische
Länder wie Deutschland oder Frankreich Dezimaltrenner und
Tausendertrenner vertauscht haben im Vergleich zu englischsprachigen
Ländern (welche die Standardspezifikationen für CSV-Dateien sind). Daher
wird in den meisten Skriptsprachen eine zusätzliche
CSV-Datei-Spezifikation read_csv2() angeboten, die explizit
für westeuropäische Länder konfiguriert ist und implizit die Spalten-
und Dezimaltrennzeichen wie im obigen Beispiel setzt.
Das Schreiben von Dateien erfolgt analog, indem einfach
write_ anstelle von read_ verwendet wird.
Die Funktionen des readr Paketes sind übersichtlich in
dessen Cheat Sheet zusammengefasst.
Frage zu CSV Import
Was ist der Unterschied zwischen read_csv() und
read_csv2()?
read_csv() und read_csv2() sind Funktionen
aus dem readr package, die beide CSV-Dateien einlesen
können. Der Unterschied liegt in der Standardkonfiguration der
Funktionen. read_csv() ist für CSV-Dateien mit Komma als
Dezimaltrenner und Punkt als Tausendertrenner konfiguriert (z.B. für
englische oder amerikanische Datenkodierungen), während
read_csv2() für CSV-Dateien mit Semikolon als
Spaltentrenner und Komma als Dezimaltrenner konfiguriert ist (wie es in
Westeuropa wie Deutschland oder Frankreich Standard ist).
Aufgabe: Datenimport
Führen sie die obigen Beispiele aus und passen sie sie an, um die
Dateien storms-2019-2021.csv und
storms-2019-2021.xlsx einzulesen. Hierzu müssen diese
Dateien ggf. zuvor gedownloaded werden (oder sie verwenden direkt die
URL zu den online verfügbaren Dateien).
Wieviele Zeilen haben die jeweiligen Datensätze? Und wie erklärt sich der Unterschied?
Die Datensätze haben unterschiedliche Größen:
-
storms-2019-2021.csvhat 19 Zeilen, es umfasst die Jahre 2019-2021. -
storms-2019-2021.xlsxhat 10 Zeilen, es umfasst nur das Jahr 2020 da nur ein einzelnes Datenblatt importiert wurde.
Pfade und Arbeitsverzeichnis
In R können Dateien relativ zum Arbeitsverzeichnis geladen
werden. Das Arbeitsverzeichnis ist der Ordner, in dem R nach Dateien
sucht, wenn keine vollständigen Pfade angegeben sind. Das
Arbeitsverzeichnis kann mit getwd() abgefragt und mit
setwd() geändert werden. In RStudio kann das
Arbeitsverzeichnis auch über das Menü “Session” -> “Set Working
Directory” -> “To Source File Location” gesetzt werden.
Dies erlaubt das Laden von Dateien ohne Angabe des vollständigen Pfades, wenn sich die Dateien im Arbeitsverzeichnis befinden. Der große Vorteil ist, dass der Code portabler wird, da er nicht mehr von absoluten Pfaden (also wwo die Daten auf ihrem Computer genau liegen) abhängt.
Alternativ können auch relative Pfade verwendet werden, um
Dateien in Unterordnern relativ zum Arbeitsverzeichnis zu laden. Zum
Beispiel, wenn sich die Datei data.csv im Unterordner
data des Arbeitsverzeichnisses befindet, kann sie mit
read_csv("data/data.csv") geladen werden.
Wenn nötig, können auch vollständige Pfade verwendet werden, um
Dateien von beliebigen Orten auf dem Computer zu laden. Das nennt man
dann einen absoluten Pfad. Ein Beispiel auf einem
Windows-System wäre
read_csv("C:/Users/Username/Documents/data.csv").
Wenn sie die letzte Pfadangabe mit der Standard-Windows-Schreibweise
vergleichen, werden sie feststellen, dass diese normalerweise mit
Backslashes \ arbeitet, also hier
"C:\Users\Username\Documents\data.csv". Das Problem ist,
dass der Backslash in R als Escape-Zeichen verwendet wird, um spezielle
TEXT-Zeichen zu kodieren. Daher wird der Backslash in Pfadangaben in R
oft verdoppelt, um dies zu umgehen. Also würde der Pfad in R so
aussehen: "C:\\Users\\Username\\Documents\\data.csv".
Alternativ können sie auch in Windows (wie im obigen Beispiel) die
“Linux-Schreibweise” verwenden, die mit Schrägstrichen /
arbeitet, die in R ohne Probleme verwendet werden können.
Falls sie unbedingt (z.B. für Copy-and-Paste Einfügen) die
Backslash-Notation der Pfade verwenden wollen, aber nicht Umformatieren
möchten, können sie den Pfad auch als raw
string codieren, in dem sie ihn statt mit Anführungszeichen
"" mit r"()" umschließen. Also in obigem
Beispiel r"(C:\Users\Username\Documents\data.csv)", wobei
sie runde durch eckige Klammern tauschen können, falls ihr Pfad auch
runde Klammern enthält.
- Dateinamen sind Textinformation und müssen in Anführungszeichen gesetzt werden.
- Pfade können absolut (eher schlecht) oder relativ zum Arbeitsverzeichnis angegeben werden. Letzteres ist portabler und empfohlen.
- Das Arbeitsverzeichnis kann mit
getwd()undsetwd()abgefragt und gesetzt werden. - In Microsoft Windows können Pfade auch mit Schrägstrichen
/statt Backslashes\geschrieben werden. - Achten sie auf die korrekte Kodierung von Textdateien, um Umlaute und Sonderzeichen korrekt einzulesen.
- Denken sie daran, dass deutsche CSV-Dateien oft Semikolon
(
;) als Trennzeichen und Komma,als Dezimaltrenner verwenden. - Excel-Dateien enthalten i.d.R. mehrere Blätter, die einzeln importiert werden müssen.
- Zusammenfassung im
readrCheat Sheet
Dieses Dokument wurde mit Unterstützung von GitHub Copilot erstellt, einem KI-gestützten Autocompletion-Tool, das auf der OpenAI GPT-3-Technologie basiert.
Content from Datenverarbeitung
Zuletzt aktualisiert am 2026-02-24 | Diese Seite bearbeiten
Geschätzte Zeit: 40 Minuten
Übersicht
Fragen
- Wie baue ich komplexe Workflows mit pipes?
- Wie transformiere ich Tabellen?
- Wie bearbeite ich Text?
- Wie führe ich mehrere Datensätze zusammen?
Ziele
- Überblick über die Möglichkeiten der Datenverarbeitung mit
tidyversePaketen
Workflows mittels Piping
In vielen Datenverarbeitungssprachen ist der Pipe-Operator ein nützliches Werkzeug, um Arbeitsschritte zu verketten, sodass das Abspeichern von Zwischenergebnisse in Variablen vermieden werden kann.
Seit R 4.1.0 ist der Pipe-Operator |> standardmäßig
in R enthalten. Alternativ bietet das magrittr package den
Pipe-Operator %>%, welcher eine etwas umfangreichere
Funktionalität bietet. Wer sich für die Unterschiede interessiert, kann
diese im Blogbetrag
dazu nachlesen. Für die meisten Anwendungen ist der
Standard-Pipe-Operator |> ausreichend und äquivalent zu
%>% verwendbar.
Der Pipe-Operator ist ein nützliches Werkzeug, um den Code lesbarer zu machen und die Arbeitsschritte in einer logischen Reihenfolge zu verketten. Hierbei wird das Ergebnis des vorherigen Arbeitsschrittes als erstes Argument des nächsten Arbeitsschrittes verwendet. Da das erste Argument von (den meisten) Funktionen der Datensatz ist, ermöglicht dies die Verknüpfung von Arbeitsschritten zu einem Workflow ohne Zwischenergebnisse speichern zu müssen oder Funktionsaufrufe zu verschachteln.
Zum Beispiel:
R
# einfaches Beispiel zur Verarbeitung eines Vektors mit base R Funktionen
# verschachtelte Funktionsaufrufe
# entspricht der Leserichtung von rechts nach links
# und einer Ergebnisbeschreibung ala "Wurzel der Summe ohne NA von 1, 2, 3, NA"
sqrt( sum( c(1, 2, 3, NA), na.rm=TRUE ) )
# mit Pipe-Operator
# entspricht der Leserichtung von links nach rechts
# und einer Beschreibung der Arbeitsschritte in ihrer Reihenfolge
# ala "nehme 1, 2, 3, NA, summiere ohne NA und davon die Wurzel"
c(1, 2, 3, NA) %>%
sum(na.rm = TRUE) %>%
sqrt()
# ok, hier nicht zwingend nötig, aber zur Veranschlaulichung des Prinzips.. ;)
Pipe-basierte workflows sind …
- intuitiv lesbar, da sie der normalen Leserichtung entsprechen.
- einfacher zu schreiben, da die umschließende Klammerung von Funktionsaufrufen entfällt. Funktionsargumente sind direkt den jeweiligen Funktionen zuordenbar.
- einfach zu erweitern, da neue Arbeitsschritte einfach an das Ende
der Kette angehängt werden können (z.B.
write_csv("bla.csv")oder bestehende Arbeitsschritte ausgetauscht werden können. - reduzieren oftmals die Anzahl der temporären Variablen, die im globalen Workspace herumliegen und die Lesbarkeit des Codes beeinträchtigen.
- einfach zu debuggen, da Zwischenergebnisse einfach ausgegeben werden
können (einfach
view()oderprint()an das Ende der entsprechenden Zeile anhängen). - einfach wiederzuverwenden, da der gesamte Workflow in einer Zeile zusammengefasst ist und immer als Block aufgerufen wird. Dadurch können keine Zwischenschritte vergessen werden und der Workflow ist immer konsistent.
Mehrzeilige R Kommandos
Wichtig: R Kommandos können über mehrere Zeilen verteilt werden, wie im obigen Beispiel zu sehen ist, was die Übersichtlichkeit des Codes erhöht.
Hierzu wird z.B. der Pipe-Operator |> ans Ende der
Zeile geschrieben und der nächste Arbeitsschritt in der nächsten Zeile
fortgesetzt. Gleiches gilt für jeden Operator (+,
==, &, ..) sowie unvollständige
Funktionsaufrufe, bei denen die Klammerung noch nicht geschlossen ist
(d.h. schließende Klammer wird in der nächsten oder einer späteren Zeile
geschrieben).
Tabellen verarbeiten
Die Transformation von Daten erfolgt i.d.R. mittels des
dplyr packages.
Grundlegend gilt: das erste Argument einer Funktion ist immer der Datensatz!
Innerhalb von dplyr Funktionen können Spaltennamen
ohne Anführungzeichen (quoting) verwendet werden.
Basistransformationen sind:
- Filtern von Zeilen mit gegebenen Kriterien (formulieren was man
BEHALTEN will!)
-
filter(storms, year == 2020)= alle Sturmdaten aus dem Jahr 2020 -
filter(storms, year == 2020 & month == 6)= alle Sturmdaten aus dem Juni 2020 -
filter(storms, !is.na(category))= alle Sturmdaten, bei denen die Kategorie bekannt ist
-
- Konkrete Zeilenauswahl (via Index oder Anzahl)
-
slice(storms, 1, 3, 5)= die Zeilen 1, 3 und 5 -
slice_tail(storms, n=10)= die 10 letzten Zeilen -
slice_max(storms, pressure)= die Zeile mit dem höchsten Wert in der Spaltepressure
-
- Sortieren von Zeilen
-
arrange(storms, year)= Sturmdaten nach Jahr aufsteigend sortieren -
arrange(storms, year, desc(month))= Sturmdaten aufsteigend nach Jahr sortieren und innerhalb eines Jahres absteigend nach Monat
-
- Duplikate entfernen
-
distinct(storms)= alle Zeilen mit identischen Werten in allen Spalten entfernen -
distinct(storms, year, month, day)= alle Zeilen mit gleichen Werten in den Spaltenyear,monthunddayentfernen (reduziert die Spalten auf die Ausgewählten) -
distinct(storms, year, month, day, .keep_all = TRUE)= alle Zeilen mit gleichen Werten in den Spaltenyear,monthunddayentfernen, aber alle Spalten behalten
-
- Auswählen/Entfernen von Spalten
-
select(storms, name, year, month, day)= nur Spalten mit Zeitinformation und Namen der Stürme behalten -
select(storms, -year, -month, -day)= Spalte mit Zeitinformation entfernen
-
- Umbenennen von Spalten
-
rename(storms, sturmname = name)= Spaltenameinsturmnameumbenennen
-
- Zusammenfassen von Daten: nur eine Zeile mit aggregierten
Informationen (z.B. Mittelwert, Summe, Anzahl, etc.) pro Gruppe
-
summarize(storms, max_wind = max(wind), num_datasets = n())= maximale Windgeschwindigkeit und Anzahl der Datensätze (Zeilen)
-
- Gruppierung von Daten = “Zerlegung” des Datensatzes in Teiltabellen,
für die anschliessende Arbeitsschritte unabängig voneinander
durchgeführt werden. Wird i.d.R. verwendet, wenn die Aufgabe “pro …”
oder “für jede …” lautet.
-
group_by(storms, year)= Gruppierung der Sturmdaten nach Jahr (aber noch keine Aggregation!) -
group_by(storms, year) |> summarize(max_wind = max(wind))= maximale Windgeschwindigkeit pro Jahr (eine “summarize” Zeile pro Teiltabelle = Gruppe = Jahr) -
group_by(storms, year) |> filter(wind == max(wind)) |> ungroup()= alle Sturmdaten, bei denen die maximale Windgeschwindigkeit pro Jahr erreicht wurde (keine Zusammenfassung!) - Grouping ist ein extrem mächtiges Werkzeug, das in vielen Situationen verwendet wird, um Daten zu transformieren. Allerdings braucht es etwas Übung, um zu verstehen, wie es funktioniert.
-
- Spalten hinzufügen/ausrechnen oder bestehende Spalten verändern
(z.B. Einheiten umrechnen)
-
mutate(storms, wind_kmh = wind * 1.60934)= Windgeschwindigkeit in km/h berechnen und als neue Spalte hinzufügen -
mutate(storms, wind = wind * 1.60934)= Windgeschwindigkeit in km/h umrechnen und bestehende Spalte überschreiben -
mutate(storms, wind_kmh = wind * 1.60934, wind_kmh_rounded = round(wind_kmh, 1))= es können auch mehrere Spalten auf einmal berechnet werden und dabei direkt neue angelegte Spalten in anschliessenden Formeln verwendet werden (hier Runden auf eine Nachkommastelle)
-
Stürme vor 1980
Erstelle eine Tabelle, welche für jeden Sturm vor 1980 neben dessen Namen nur das Jahr und dessen Status beinhaltet und nach Jahr und Status sortiert ist.
Verwenden sie eine Pipe die folgende Funktionen verbindet -
filter() - select() - distinct()
- arrange()
R
# Ausgangsdatensatz = Beginn der Pipe
storms |>
# Zeilen filtern
filter(year < 1980) |>
# Spalten auswählen
select(name, year, status) |>
# doppelte Zeilen entfernen
distinct() |>
# sortieren
arrange(year, status)
AUSGABE
# A tibble: 129 × 3
name year status
<chr> <dbl> <fct>
1 Amy 1975 extratropical
2 Blanche 1975 extratropical
3 Doris 1975 extratropical
4 Eloise 1975 extratropical
5 Gladys 1975 extratropical
6 Hallie 1975 extratropical
7 Blanche 1975 hurricane
8 Caroline 1975 hurricane
9 Doris 1975 hurricane
10 Eloise 1975 hurricane
# ℹ 119 more rowsGruppieren und Aggregieren
Eine der wichtigsten Funktionen in dplyr ist das
Gruppieren von Daten und das Aggregieren von Werten innerhalb dieser
Gruppen. Dies wird in der Regel mit den Funktionen
group_by() und summarize() durchgeführt.
group_by() teilt den Datensatz in Gruppen (imaginäre
Teiltabellen) auf, basierend auf den Werten in einer oder mehreren
Spalten. Im Anschluß wird, vereinfacht gesagt, für jede Gruppe eine
separate Berechnung durchgeführt.
Die Funktion summarize() ist die am häufigsten
verwendete Funktion, um Werte innerhalb dieser Gruppen zu
aggregieren.
Ein einfaches Beispiel:
R
storms |>
# Tabelle nach Jahr gruppieren
group_by(year) |>
# maximale Windgeschwindigkeit pro Jahr berechnen
summarize(max_wind = max(wind))
AUSGABE
# A tibble: 50 × 2
year max_wind
<dbl> <int>
1 1975 120
2 1976 105
3 1977 150
4 1978 120
5 1979 150
6 1980 165
7 1981 115
8 1982 115
9 1983 100
10 1984 115
# ℹ 40 more rowsIn diesem Beispiel wird die Tabelle storms nach dem Jahr
gruppiert und für jede Gruppe (d.h. jedes Jahr) die maximale
Windgeschwindigkeit berechnet. Das Ergebnis ist eine Tabelle mit zwei
Spalten: year und max_wind.
Achtung
Zu beachten ist, dass Gruppierungen in dplyr nur
virtuell sind und nicht zu einer physischen Aufteilung des Datensatzes
führen. Allerdings wird diese Gruppierung aufrecht erhalten, bis sie
explizit aufgehoben wird (z.B. durch ungroup()) oder dies
implizit durch eine andere Funktion geschieht (z.B. “schließt”
summarize() die letzte Gruppierung).
Gruppiertes Filtern
Erstelle eine Tabelle, welche für jeden Sturmstatus das Jahr und den Namen des letzten Sturms auflistet.
AUSGABE
# A tibble: 9 × 3
status year name
<fct> <dbl> <chr>
1 tropical wave 2018 Kirk
2 subtropical depression 2023 Don
3 extratropical 2024 Patty
4 subtropical storm 2024 Patty
5 hurricane 2024 Rafael
6 other low 2024 Rafael
7 disturbance 2024 Sara
8 tropical storm 2024 Sara
9 tropical depression 2024 Sara - Gruppieren sie die Tabellendaten nach
status, um für jeden Sturmtyp eine “Teiltabelle” zu erhalten - Um den letzten Sturm zu finden
- Möglichkeit 1: neue Spalte mit Zeitinformation zusammensetzen und damit die Zeile mit maximalen Datum (pro Teiltabelle) extrahieren
- Möglichkeit 2: Teiltabellen bzgl. Zeitspalten sortieren und erste bzw. letzte Zeile extrahieren
Alternative 1
R
storms |>
# decompose table by storm status
group_by(status) |>
# encode time information of each entry in a single column
mutate(date = parse_date_time(str_c(year,month,day,hour,sep="-"), "%Y-%m-%d-%H")) |>
# filter for the latest date
filter(date == max(date)) |>
# ALTERNATIVELY cut out row with latest date
# slice_max(date, n=1) |>
# rejoin table information and undo grouping
ungroup() |>
select(status, year, name)
Alternative 2
R
storms |>
# decompose table by storm status
group_by(status) |>
# sort ascending by date (hierarchical sorting)
arrange(year,month,day,hour) |>
# pick last row w.r.t. sorting
slice_tail(n=1) |>
# ALTERNATIVELY pick row with last index
# slice( n() ) |>
# OR do both (sort+pick) directly with slice_max
# slice_max( tibble(year,month,day,hour), n=1, with_ties = F ) |>
# rejoin table information and undo grouping
ungroup() |>
select(status, year, name)
Eine Zusammenfassung der wichtigsten Funktionen in dplyr
finden sich in dessen Cheat Sheet.
Tabellengestalt ändern
Die “tidy” Form ist eine spezielle Form von tabellarischen Daten, die für viele Datenverarbeitungsschritte geeignet ist. In der “tidy” Form sind die Daten so organisiert, dass jede Beobachtung (z.B. Messung) in einer Zeile notiert ist und jede Variable (also z.B. Beschreibungungen und Messwerte) eine Spalte definieren. Diese Art der Tabellenform wird auch “long table” oder “schmal” genannt, weil die Daten in einer langen, schmalen Tabelle organisiert sind.
Allerdings werden Rohdaten häufig in einer “ungünstigen” Form vorliegen, die für die weitere Verarbeitung nicht optimal ist. Manuelle Datenerfassung erfolgt oft in grafischen Oberflächen wie MS Excel, worin die Daten i.d.R. in einer “breiten” oder “wide table” Form gespeichert, in der eine Variable (z.B. Messwert) in mehreren Spalten gespeichert ist. In der “breiten” Form sind die Daten so organisiert, dass Beobachtungen (und ihre Messwerte) in Spalten gruppiert sind, während die Variablen in den Zeilen stehen.
Hier ein Beispiel aus dem Tutorial Introduction to R/tidyverse for Exploratory Data Analysis

Das obige Beispiel zeigt eine “breite” Tabelle (rechts) und eine
“lange” Tabelle (links) mit den gleichen Daten. Die Transformation
zwischen beiden Formaten kann durch die Funktionen
pivot_longer() und pivot_wider() erreicht
werden. Beachten sie im Beispiel, dass die Spaltennamen des wide table
Formats in beide Transformationsrichtungen verarbeitet werden
können.
Vor- und Nachteile
Während das wide table Format kompakter und damit ggf. besser zur Datenerfassung und -übersicht geeignet ist, ist das long table Format besser für die Datenanalyse und -visualisierung geeignet. In letzterem liegt Information redundant vor, da in jeder Zeile die komplette Information einer Beobachtung vorliegen muss.
Die tidyverse Bibliothek tidyr bietet Funktionen, um
Daten zwischen diesen beiden Formen zu transformieren. Dies wird auch
als “reshaping” oder “pivotieren” bezeichnet. Hiermit können Spalten in
Zeilen und umgekehrt umgeformt werden.
Das Beispiel hierfür ist etwas länger, um für Anschauungszwecke die Datentabelle erst etwas einzukürzen.
R
storms |>
filter(name == "Arthur" & year == 2020) |> # speziellen Sturm auswählen
select(name, year, month, day, wind, pressure) |> # (zur Vereinfachung) nur spezifische Spalten
# Verteile Wind und Druck in separate Zeilen mit entsprechenden Labels in einer neuen Spalte "measure"
pivot_longer(cols = c(wind, pressure), names_to = "measure", values_to = "value") |>
slice_head(n=4) # nur die ersten 4 Zeilen anzeigen
AUSGABE
# A tibble: 4 × 6
name year month day measure value
<chr> <dbl> <dbl> <int> <chr> <int>
1 Arthur 2020 5 16 wind 30
2 Arthur 2020 5 16 pressure 1008
3 Arthur 2020 5 17 wind 35
4 Arthur 2020 5 17 pressure 1006Details und weitere Funktionen und Beispiele sind im Cheat Sheets des
tidyr Paketes übersichtlich zusammengefasst.
Textdaten verändern und verwenden
Um Textdaten (Strings) zu verändern, gibt es verschiedene
Funktionen des stringr Paketes, die in Kombination mit
mutate() oder anderen Funktionen verwendet werden können.
Die meisten Funktionen des stringr Paketes sind sehr
intuitiv und haben eine ähnliche Syntax wie die Funktionen des
dplyr Paketes. Zudem sind viele mit regulären Ausdrücken
verwendbar, um Textdaten aufgrund von allgemeineren Textmustern zu
erkennen und zu verändern. Alle Funktionen des stringr
Paketes beginnen mit str_ und sind in der Dokumentation
aufgeführt. Einige wichtige sind:
-
str_c(): Verkettet mehrere Strings zu einem, analog topaste0() -
str_detect(): Prüft, ob ein String einen bestimmten Textteil enthält -
str_replace(): Ersetzt einen Teil eines Strings durch einen anderen -
str_to_lower()/str_to_upper: Wandelt alle Buchstaben in Klein-/Grossbuchstaben um -
str_sub(): Extrahiert einen Teil eines Strings -
str_trim(): Entfernt Leerzeichen am Anfang und Ende eines Strings -
str_extract(): Extrahiert einen Teil eines Strings, der einem bestimmten Muster entspricht -
str_remove(): Entfernt einen Teil eines Strings, der einem bestimmten Muster entspricht -
str_split(): Teilt einen String an einem bestimmten Trennzeichen auf
Die meisten Funktionen liefern nur einen Treffer oder verändern nur
den ersten Treffer. Daher gibt es von vielen Funktionen Varianten, die
alle Treffer finden und/oder verarbeiten, z.B.
str_detect_all(), str_replace_all(),
str_extract_all().
Die Funktionen sind im stringr
Cheat Sheet zusammengefasst.
Beispiele für deren Verwendung mit mutate() und
Co. sind:
R
# Beispiel: Grossbuchstaben in Spalte "name" in Kleinbuchstaben umwandeln
# (und nur eindeutige Einträge in Spalte "name" anzeigen)
mutate(storms, name = str_to_lower(name)) |> distinct(name)
# Beispiel: Jahrhundert aus Spalte "year" extrahieren
mutate(storms, century = str_sub(year, 1, 2)) |> distinct(name, century)
# oder via regulärem Ausdruck
mutate(storms, century = str_extract(year, "^\\d{2}"))
mutate(storms, century = str_remove(year, "..$"))
# Beispiel: Nur Stürme mit "storm" im Status behalten
filter(storms, str_detect(status, "storm")) |> distinct(name)
Beachte:
- wenn die (Daten)Eingabe einer
stringrFunktion ein Vektor ist, wird die Funktion auf jedes Element des Vektors angewendet und gibt einen Vektor mit den Ergebnissen zurück. (= vektorisierte Prozessierung = zentrales Verarbeitungsprinzip in R) - wenn die Eingabe kein String ist (z.B. eine Zahl), wird die Eingabe in einen String umgewandelt und die Funktion auf den entstandenen String angewendet. (= automatische Typumwandlung, sogenanntes coercion in R)
Reguläre Ausdrücke
Für komplexe Textverarbeitung werden oft reguläre Ausdrücke verwendet. Diese erlauben es, Textmuster zu definieren, die in einem Text gesucht, ersetzt oder extrahiert werden können.
Für eine Einführung in reguläre Ausdrücke siehe RegexOne Tutorial oder RStudio RegEx Cheat Sheet (pdf).
Reguläre Ausdrücke
Betrachten sie noch einmal die beiden mutate() Beispiele
von oben, in denen das Jahrhundert aus der Spalte “year” mit Hilfe von
regulären Ausdrücken extrahiert wird.
Was genau definieren die beiden regulären Ausdrücke
^\\d{2} und ..$?
-
^\\d{2}= “die ersten zwei ({2}) Ziffern (\\d) am Textanfang (^)”- hier genutzt um das Jahrhundert mit
str_extract()zu extrahieren
- hier genutzt um das Jahrhundert mit
-
..$= “die letzten zwei beliebigen Zeichen (..) am Ende des Textes ($)”- hier genutzt um die letzten zwei Zeichen (Jahrzehnt) mit
str_remove()zu entfernen
- hier genutzt um die letzten zwei Zeichen (Jahrzehnt) mit
Daten zusammenführen
Oftmals liegen daten in mehreren Tabellen vor, die zusammengeführt werden müssen, um die Daten zu analysieren. Zum Beispiel können Daten in einer Tabelle die Anzahl der Sturmtage pro Jahr enthalten und in einer anderen Tabelle die Kosten für Sturmschäden pro Jahr.
R
# Datensatz mit Sturmschäden pro Jahr (zufällige Werte) für 2015-2024
costs <-
tibble(year = 2015:2024, # Jahresbereich festlegen
costs = runif(length(year), 1e6, 1e8)) # Zufallsdaten gleichverteilt erzeugen
# Anzahl der Sturmtage pro Jahr
stormyDays <-
storms |>
# Datensatz in Einzeljahre aufteilen
group_by(year) |>
# nur eine Zeile pro Monat/Jahr Kombination (pro Jahr) behalten
distinct(month, day) |>
# zählen, wie viele Zeilen pro Jahr noch vorhanden sind
count() |>
ungroup()
# Beispiel (1): Sturmtaginformation (links) mit Kosteninformation (rechts) ergänzen
# BEACHTE: für Jahre ohne Eintrag in `costs` wird `NA` eingetragen !
left_join(stormyDays, costs, by = "year") |>
# nur die ersten und letzten 3 Zeilen anzeigen
slice( c(1:3, (n()-2):n()) )
# Beispiel (2): nur Datensätze für die beides, d.h. Sturmtage als auch Kosten, vorhanden ist
inner_join(stormyDays, costs, by = "year")
Spaltennamen
Studieren sie die Hilfeseite der join Funktionen und finden sie heraus, wie sie die Spaltennamen der beiden Tabellen angeben können, um die Daten zu verbinden, wenn die gemeinsamen Daten in unterschiedlich benannten Spalten stehen.
Das mapping der Spalten erfolgt über einen benannten Vektor, d.h. die Spaltennamen der linken Tabelle werden als Elementnamen für die entsprechenden Spaltennamen der rechten Tabelle verwendet:
R
stormyDays |>
rename( Jahr = year ) |> # Jahresspalte exemplarisch umbenannt
filter( Jahr >= 2013 ) |>
# geänderte Sturmtaginformation (links) aus der pipe
# mit Kosteninformation (rechts) aus expliziter Tabelle "costs" ergänzen
left_join( costs,
# Explizites mapping von costs$year auf die "Jahr" Spalte der linken Tabelle
# Beachten sie das quoting der Spaltennamen!
by = c(Jahr = "year")
# Alternativ kann das mapping auch über eine Funktion erfolgen:
# Hier sind keine Anführungszeichen notwendig:
# by = join_by(Jahr = year)
)
- Speichern sie Daten nur in Variablen zwischen, wenn sie diese Daten mehrfach benötigen.
- Verwenden sie Pipes (
|>) um Daten durch eine Reihe von Transformationen zu leiten. - Versuchen sie die Datenverarbeitung in kleine, leicht verständliche Schritte zu unterteilen.
- Vermeiden sie unnötige Schleifen und Schachtelungen, das meiste lässt sich mit Grouping, vektorisierten Operationen und Joins kompakter und eleganter lösen.
- Auch explizite Elementzugriffe (z.B.
df$col) und -operationen können i.d.R. effizient durchdplyrFunktionen ersetzt werden. - Zusammenfassungen der Pakete:
Dieses Dokument wurde mit Unterstützung von GitHub Copilot erstellt, einem KI-gestützten Autocompletion-Tool, das auf der OpenAI GPT-3-Technologie basiert.
Content from Visualisierung
Zuletzt aktualisiert am 2026-02-24 | Diese Seite bearbeiten
Geschätzte Zeit: 25 Minuten
Übersicht
Fragen
- Wie visualisiere ich Daten?
Ziele
- Datenvisualisierung mit
ggplot2
Die Visualisierung von Daten ist ein wichtiger Bestandteil der
Datenanalyse, da sie es ermöglicht, Muster und Zusammenhänge in den
Daten zu erkennen und zu kommunizieren. In R wird die Visualisierung von
Daten mit dem ggplot2 Paket durchgeführt, das auf der Grammar of
Graphics basiert. Hierbei müssen die Daten in tabellarischer
Form vorliegen, d.h. jede Zeile entspricht einem Datensatz und jede
Spalte einer Variable (“tidy data”).
Die Visualisierung von Daten wird in verschiedene Schichten (z.B.
Punkte, Linien, Balken) und Eigenschaften (z.B. x-Achse, y-Achse, Farbe,
Form) unterteilt. Die Verküpfung von Tabellenspalten mit den Ebenen und
Eigenschaften (d.h. Welche Information wird wie fürs Plotting verwendet)
erfolgt über das Argument mapping = und die
aes() Funktion. Schichten (z.B. Balken, Linien, Punkte)
werden mit geom_*() Funktionen hinzugefügt, wobei die Daten
für die Schicht über das Argument data = übergeben
werden.
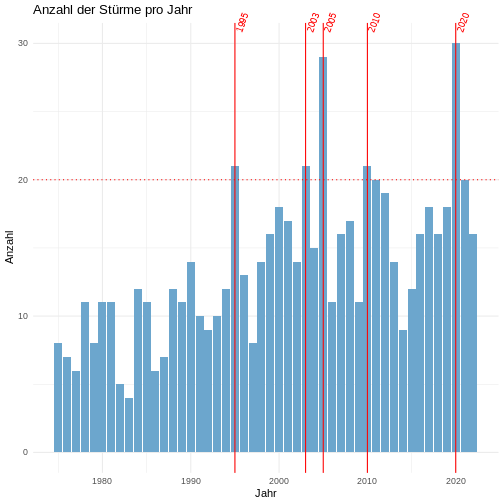
Im Folgenden wird ein umfangreiches Beispiel für die Visualisierung
von Daten mit ggplot2 gezeigt, bei dem die Anzahl der
Stürme pro Jahr visualisiert wird.
R
# Beispiel: Anzahl der Stürme pro Jahr visualisieren
# Rohdatensatz startet Visualisierungsworkflow
storms |>
########### Datenvorbereitung #############
# auf einen Eintrag (Zeile) pro Sturm und Jahr reduzieren
distinct(year, name) |>
# Anzahl der Stürme pro Jahr zählen
group_by(year) |>
count() |>
ungroup() |>
########### Datenvisualisierung #############
# ggplot-Objekt erstellen (Daten via pipe übergeben)
ggplot(mapping = aes(x = year, y=n)) + # Verknüpfung von Datenspalten (year, n) und Achsen (x,y)
# Diagrammtitel und Achsenbeschriftung
labs(title = "Anzahl der Stürme pro Jahr", x = "Jahr", y = "Anzahl") +
# grundlegende Diagrammformatierung (Hintergrundfarben, Schriftarten, ...)
theme_minimal() +
# Balkendiagramm mit Anzahl der Stürme pro Jahr
geom_bar(fill="skyblue3", stat = "identity") +
# gepunktete horizontale Linie bei 20
geom_hline(yintercept = 20, linetype="dotted", col="red") +
# Highlighting der Jahre mit mehr als 20 Stürmen
geom_vline(data = ~ filter(.x, n>20), # Datensatz einschränken
# (hier `.x` = Platzhalter für Daten aus vorheriger Ebene, d.h. `storms`)
aes(xintercept=year), # welche (Teil)Tabellendaten für Position zu verwenden
color="red") + # Zusätzliche Formatierung
# Jahreszahlen der Jahre mit mehr als 20 Stürmen in schräger Textausrichtung
geom_text(data = ~ filter(.x, n>20), # Datensatz einschränken
aes(label=year, x=year, y=max(n)), # Daten und Positionen festlegen
angle=70, vjust=0.5, hjust=-0.6, size=3, color="red") + # Textformatierung
# disable clipping um Jahreszahltexte außerhalb des Diagrammbereichs anzuzeigen
coord_cartesian(clip = "off")
R
# optional: (zuletzt gemaltes) Diagramm speichern
ggsave("storms_per_year.png", width=10, height=5, dpi=300)
Als Nachschlagewerk empfiehlt sich das offizielle Cheat Sheet des
ggplot2 Pakets, welches die wichtigsten Funktionen und
Argumente übersichtlich darstellt:
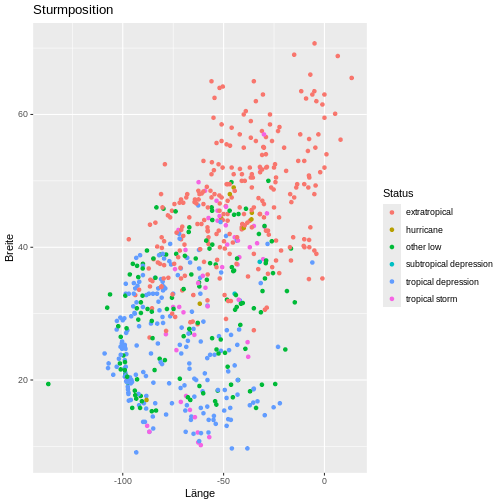
Sturmposition visualisieren
Zeichnen sie ein Punktdiagram, welches für folgenden Datensatz
- Länge (x-Achse) und Breite (y-Achse) der Messung zeigt und die Punkte anhand des Sturmstatus einfärbt,
- die Achsen mit “Länge” und “Breite” beschriftet, und
- den Diagrammtitel “Sturmposition” hat.
R
storms |>
group_by(name,year) |>
slice_tail(n=1) |>
ggplot(mapping = aes(x = long, y = lat, color = status)) +
geom_point() +
labs(
title="Sturmposition",
x="Länge",
y="Breite",
color="Status" # ansonsten klein geschrieben
)
R
# alternatively
# geom_title("Sturmposition") + geom_xlab("Länge") + geom_ylab("Breite") + geom_color("Status")
-
ggplot2benötigt einendata.frameals Eingabe, welcher “tidy” ist (d.h. eine Zeile pro Beobachtung und eine Spalte pro Variable). - Das
mappingArgument ermöglicht mittels deraes()Funktion die Verknüpfung von Variablen des Datensatzes (d.h. Spaltennamen) mit visuellen Eigenschaften (z.B. x-Achse, Farbe, Größe). -
geom_*Funktionen fügen dem Plot Schichten hinzu (z.B. Punkte, Linien, Balken). -
labs()ermöglicht die Anpassung von Diagrammtitel und Achsenbeschriftung. -
theme_*Funktionen ermöglichen die Anpassung genereller Diagrammformatierungen (z.B. Hintergrundfarben, Schriftarten). - Es gibt viele weitere Funktionen und Argumente, um die Darstellung
von Diagrammen zu verfeinern (z.B.
facet_wrap(),scale_*,coord_*). - Diagramme mit
ggsave()in beliebigem Dateiformat (PNG, PDF, SVG, ..) speichern. - Ausserdem gibt es viele Erweiterungen für
ggplot2(z.B.ggplotly,ggrepel), die zusätzliche Funktionalitäten oder Visualisierungstypen/-diagramme bieten. - Zusammenfassung im
ggplot2Cheat Sheet
Dieses Dokument wurde mit Unterstützung von GitHub Copilot erstellt, einem KI-gestützten Autocompletion-Tool, das auf der OpenAI GPT-3-Technologie basiert.





